DBSCANは、密度に基づいてデータをグループ化するアルゴリズムで、ノイズを含むデータにも強い特徴があります。
製造業では、センサーから得られる振動データをDBSCANで分析し、異常な振る舞いを早期検知するなど、異常検知や設備の故障予兆の分析、品質管理において活用されています。
この記事では、製造現場でのDBSCANの活用例を紹介し、アルゴリズムの基本的な仕組みや実装方法を解説します。
すぐに実践したい方のために、コピペで使える自作クラスも公開していますので、ぜひご一読ください。
DBSCANとは
DBSCAN(Density-Based Spatial Clustering of Applications with Noise)は、密度に基づいたクラスタリング手法の一つで、データポイントを密度の高い領域(クラスター)とそれ以外の領域(ノイズ)に分類するアルゴリズムです。1996年にMartin Esterらによって提案されました。
アルゴリズムの概要
- 初期化
DBSCANはデータセット内の各ポイントを未分類の状態から始めます。 - コアポイントの識別
各データポイントについて、周囲epsの範囲内に存在するポイントの数を数えます。この数がmin_samples以上であれば、そのポイントはコアポイントと見なされます。 - クラスタの形成
コアポイントから開始し、そのeps範囲内にあるすべてのポイントを同じクラスタに割り当てます。これにより、新たに見つかったコアポイントに対しても同様の処理を繰り返し、クラスタを拡張します。 - 境界ポイントの分類
コアポイントのeps範囲内にあるが、他のコアポイントの数が少ないデータポイントは境界ポイントと見なされ、コアポイントが属するクラスタに割り当てられます。 - ノイズの識別
クラスタに属さないポイント(周囲のポイント数がmin_samples未満のもの)はノイズとして識別され、-1のラベルが付けられます。
DBSCANのメリット
- 形状が不規則なクラスターも検出可能
球状に限定されず、複雑で非線形な形状のクラスターを識別できます。 - ノイズや外れ値を自然に扱える
明確にノイズポイントとして分類できるため、外れ値検出にも適しています。 - クラスター数を事前に指定する必要がない
データの密度に基づいてクラスターを動的に形成するため、クラスター数を事前に決める必要がありません。
DBSCANの課題
- パラメータ(εとMinPts)の設定が結果に大きく影響する
適切なパラメータを見つけるのが難しく、結果が極端に変わる場合があります。 - 高次元データや非常に異なるスケールのデータには不向き
次元が増えると距離計算が不安定になり、クラスタリング精度が低下します。 - εの範囲が小さい場合、データ全体がノイズとみなされる可能性がある
小さすぎるεはクラスター形成を妨げ、大部分のデータがノイズ扱いになることがあります。
クラスタリングに必要なモジュールのインストール
クラスタリングを実装するには、Pythonとscikit-learnライブラリが必要です。以下のコマンドで必要なライブラリをインストールできます。
pip install scikit-learn
pip install numpy
pip install pandas
pip install matplotlib
DBSCANを使ったクラスタリングの実装例
DBSCANでは、スケーリングが異なるデータや、高次元データの分類が苦手です。
必要に応じて、事前に標準化や正規化でスケールを整えたり、主成分分析などで次元削減を行うなどの前処理を行うと効果的です。
DBSCAN(Density-Based Spatial Clustering of Applications with Noise)は、密度に基づいたクラスタリング手法です。異常検知に適しており、クラスタリングできない外れ値を「ノイズ」として扱い、-1 を返します。DBSCANでは、クラスタの形成に必要なパラメータとして、データポイントが同じクラスタであると見なす最大距離(eps)、クラスタと見なすための最小ポイント数(min_samples)があります。
例えば、eps=1.0とmin_samples=5の場合、各データポイントの周り1.0の距離以内に少なくとも5つのデータポイントが存在する場合に、そのポイントはクラスタの一部と見なされます。これにより、密度の高い領域がクラスタとして認識され、密度の低い領域はノイズとして分類されます。
これらのパラメータはデータセットによって調整が必要で、適切に設定することで効果的なクラスタリングと異常検知が可能となります。
from sklearn.cluster import DBSCAN
# 5個の説明変数を持つ6組分のテストデータを生成
data = [
[5.1, 3.5, 1.4, 0.2, 0.3],
[4.9, 3.0, 1.4, 0.2, 0.4],
[4.7, 3.2, 1.3, 0.2, 0.3],
[4.5, 3.1, 1.5, 0.2, 0.3],
[5.0, 3.6, 1.4, 0.2, 0.4],
[4.6, 3.1, 1.3, 0.2, 0.3]
]
# DBSCANクラスタリングの実行
dbscan = DBSCAN(eps=0.3, min_samples=2)
labels = dbscan.fit_predict(data)
core_sample_indices = dbscan.core_sample_indices_
print("====分類結果====")
print(labels)
print("====コアサンプルのインデックス====")
print(core_sample_indices)下記は分類結果です。2番目のデータはノイズとして認識され、それ以外は0と1に分類されていることが分かります。
コアサンプルのインデックスは、各クラスターのコアとなるデータポイントのインデックス番号になります。
====分類結果====
[ 0 -1 1 1 0 1]
====コアサンプルのインデックス====
[0 2 3 4 5]
パラメータ設定について
| パラメータ | 説明 |
|---|---|
| eps(ε) | データポイントが同じクラスタに属すると見なす最大距離。この距離以内に他のデータポイントが存在する場合、そのデータポイントは同じクラスタに属します。 この値クラスタの感度に大きく影響し、クラスタの感度に大きく影響します値が小さすぎるとクラスタが分裂しすぎ、大きすぎると全てが一つのクラスタになりがちです。データの分布を見ながら適切な値を設定します。 |
| min_samples | あるデータポイントをコアポイントと見なすために必要な最小データポイント数。この数以上のポイントがepsの範囲内に存在する場合、そのポイントはコアポイントとなります。 |
最適なepsを見つける
epsがクラスタリングの結果を大きく左右しますが、どのような値を試せばいいのでしょうか?
この章では、最適なepsを求めるための方法について紹介します。
k距離プロット(エルボー法)
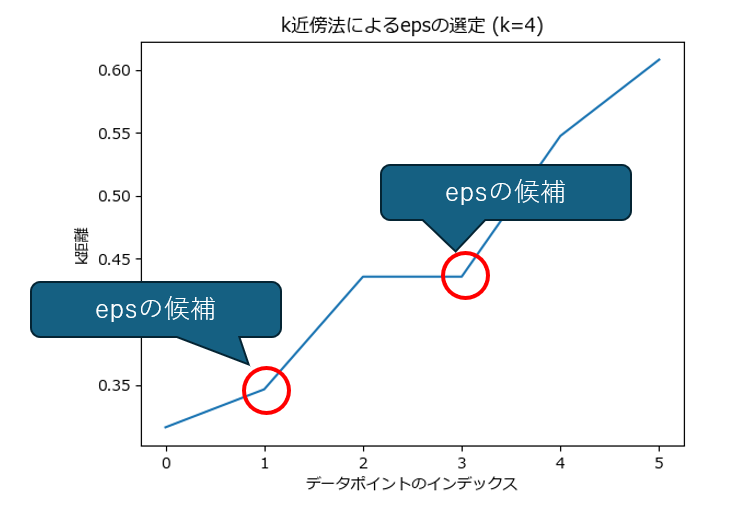
k近傍法を使ってデータポイント間の距離分布を可視化し、屈曲点(エルボーポイント)からepsを見つける方法です。
屈曲点より左側のデータポイントは高密度領域に属しており、クラスタの中にあると考えられます。
屈曲点より右側のデータポイントは、疎な領域や外れ値に属している可能性が高いと考えられます。
グラフの形状から、急激に距離が増加し始める点を探すことで、epsの候補を見つけることができます。
K は4から始め、満足する結果が得られるまで~10程度を試すのが一般的です。
実際の最適値は、見つかった候補(上図では1と3)の前後に存在するため、トライ&エラーで最適値を探す必要があります。
import numpy as np
from sklearn.neighbors import NearestNeighbors
import matplotlib.pyplot as plt
from matplotlib import rcParams
rcParams['font.family'] = 'Meiryo'
# サンプルデータ
data = [
[5.1, 3.5, 1.4, 0.2, 0.3],
[4.9, 3.0, 1.4, 0.2, 0.4],
[4.7, 3.2, 1.3, 0.2, 0.3],
[4.5, 3.1, 1.5, 0.2, 0.3],
[5.0, 3.6, 1.4, 0.2, 0.4],
[4.6, 3.1, 1.3, 0.2, 0.3]
]
# k距離プロット
def plot_k_distance(data, k=4):
nn = NearestNeighbors(n_neighbors=k)
nn.fit(data)
distances, _ = nn.kneighbors(data)
k_distances = np.sort(distances[:, k - 1])
plt.plot(k_distances)
plt.xlabel("データポイントのインデックス")
plt.ylabel("k距離")
plt.title(f"k近傍法によるepsの選定 (k={k})")
plt.show()
plot_k_distance(data, k=4)
シルエットスコア(Silhouette Score)
シルエットスコアは、クラスタ内の一貫性とクラスタ間の分離を評価する指標です。DBSCAN のクラスタリングに対して異なる eps 値を試してシルエットスコアを計算し、最も高いスコアを示す eps を選ぶ方法です。
シルエットスコアはクラスタリングの評価指標として有用ですが、必ずしも必須ではありません。特に、DBSCANのような密度ベースのアルゴリズムでは、クラスタリングの結果が必ずしも明確な境界を持つとは限らないため、シルエットスコアが低い場合もあります。そのため、シルエットスコアは参考程度と考えてください。
import numpy as np
from sklearn.cluster import DBSCAN
from sklearn.model_selection import GridSearchCV
import matplotlib.pyplot as plt
# サンプルデータ
data = np.array([
[5.1, 3.5, 1.4, 0.2, 0.3],
[4.9, 3.0, 1.4, 0.2, 0.4],
[4.7, 3.2, 1.3, 0.2, 0.3],
[4.5, 3.1, 1.5, 0.2, 0.3],
[5.0, 3.6, 1.4, 0.2, 0.4],
[4.6, 3.1, 1.3, 0.2, 0.3]
])
from sklearn.cluster import DBSCAN
from sklearn.metrics import silhouette_score
import numpy as np
eps_values = np.linspace(0.1, 10.0, 1000) # eps 値の範囲
best_eps = 0
best_score = -1
for eps in eps_values:
db = DBSCAN(eps=eps)
labels = db.fit_predict(data)
if len(set(labels)) > 1: # クラスタが2つ以上ある場合
score = silhouette_score(data, labels)
if score > best_score:
best_score = score
best_eps = eps
print(f"最適なeps: {best_eps}, シルエットスコア: {best_score}")最適なeps: 0.5162162162162163, シルエットスコア: 0.1491456447147945
- スコアが 1 に近いほど、クラスタが適切に分けられていることを示します(良い結果)。
- スコアが 0 に近い場合、データポイントがどのクラスタにも適切に属していない可能性があります。
- スコアが 負の値 に近いと、データポイントが誤ったクラスタに分類されている可能性があります。
| シルエットスコアが高い (例えば、0.5以上) | クラスタリングが適切に行われた可能性が高い |
|---|---|
| シルエットスコアが低い (例えば、0.2以下) | クラスタリング結果があまり良くない可能性がある。 密度のしきい値 eps を調整することで改善できる場合もあるが、そもそもクラスタリングの設定(min_samples や eps)がデータに合っていない可能性もある。 |
可視化
可視化は、クラスタリング対象のデータを単純に散布図でプロットするのですが、クラスタリング結果(番号)に応じて色分けをしています。
また、ここでは2次元、3次元データの可視化方法と合わせて、多次元データを次元削減して可視化する具体的な方法も紹介します。
2次元データのクラスタリング結果を可視化
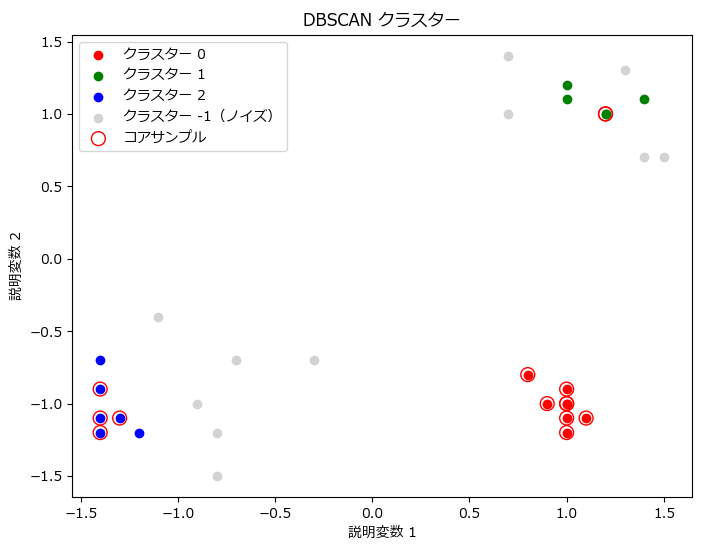
from sklearn.cluster import DBSCAN
import numpy as np
import matplotlib.pyplot as plt
from matplotlib import rcParams
rcParams['font.family'] = 'Meiryo'
# ダミーデータの生成
data = np.array([
[1.5, 0.7],[1.0, -1.1],[1.0, 1.1],[-1.4, -0.7],[-1.2, -1.2],
[1.2, 1.0],[0.7, 1.4],[1.0, -1.0],[1.0, -1.2],[1.3, 1.3],
[-1.4, -0.9],[-0.3, -0.7],[-1.4, -1.2],[1.4, 0.7],[-0.9, -1.0],
[-1.3, -1.1],[0.7, 1.0],[0.8, -0.8],[1.0, 1.2],[-0.7, -0.7],
[-1.4, -1.1],[1.4, 1.1],[-1.1, -0.4],[0.9, -1.0],[1.0, -1.0],
[1.1, -1.1],[-0.8, -1.5],[1.0, -0.9],[-0.8, -1.2],[1.2, 1.0],
])
# DBSCANクラスタリングの実行
dbscan = DBSCAN(eps=0.3, min_samples=5)
labels = dbscan.fit_predict(data)
core_sample_indices = dbscan.core_sample_indices_
print("====分類結果====")
print(labels)
# クラスタリング結果の可視化
plt.figure(figsize=(8, 6))
unique_labels = set(labels)
colors = ['r', 'g', 'b', 'c', 'm', 'y', 'k']
for k, col in zip(unique_labels, colors):
label = f'クラスター {k}'
if k == -1:
# ノイズポイントは薄いグレーで表示
col = 'lightgray'
label += '(ノイズ)'
class_member_mask = (labels == k)
xy = data[class_member_mask]
plt.scatter(xy[:, 0], xy[:, 1], c=[col], label=label)
# コアサンプルを赤でプロット
core_samples = data[core_sample_indices]
plt.scatter(core_samples[:, 0], core_samples[:, 1], s=100, facecolors='none', edgecolors='r', label='コアサンプル')
plt.title('DBSCAN クラスター')
plt.xlabel('説明変数 1')
plt.ylabel('説明変数 2')
plt.legend()
plt.show()
3次元データのクラスタリング結果を可視化
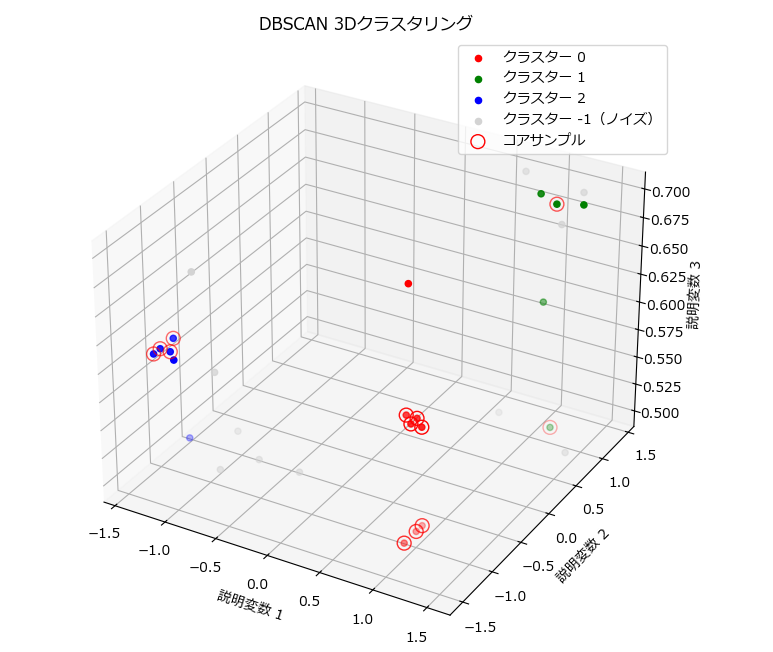
from sklearn.cluster import DBSCAN
import numpy as np
import matplotlib.pyplot as plt
from matplotlib import rcParams
from mpl_toolkits.mplot3d import Axes3D
rcParams['font.family'] = 'Meiryo'
# ダミーデータの生成
data = np.array([
[1.5, 0.7, 0.5], [1.0, -1.1, 0.6], [1.0, 1.1, 0.7], [-1.4, -0.7, 0.5], [-1.2, -1.2, 0.6],
[1.2, 1.0, 0.5], [0.7, 1.4, 0.7], [1.0, -1.0, 0.6], [1.0, -1.2, 0.5], [1.3, 1.3, 0.7],
[-1.4, -0.9, 0.6], [-0.3, -0.7, 0.5], [-1.4, -1.2, 0.6], [1.4, 0.7, 0.7], [-0.9, -1.0, 0.5],
[-1.3, -1.1, 0.6], [0.7, 1.0, 0.5], [0.8, -0.8, 0.7], [1.0, 1.2, 0.6], [-0.7, -0.7, 0.5],
[-1.4, -1.1, 0.6], [1.4, 1.1, 0.7], [-1.1, -0.4, 0.5], [0.9, -1.0, 0.6], [1.0, -1.0, 0.5],
[1.1, -1.1, 0.6], [-0.8, -1.5, 0.7], [1.0, -0.9, 0.5], [-0.8, -1.2, 0.6], [1.2, 1.0, 0.7]
])
# DBSCANクラスタリングの実行
dbscan = DBSCAN(eps=0.3, min_samples=5)
labels = dbscan.fit_predict(data)
core_sample_indices = dbscan.core_sample_indices_
print("====分類結果====")
print(labels)
print("====コアサンプル====")
print(data[core_sample_indices])
# クラスタリング結果の可視化(3D)
fig = plt.figure(figsize=(10, 8))
ax = fig.add_subplot(111, projection='3d')
unique_labels = set(labels)
colors = ['r', 'g', 'b', 'c', 'm', 'y', 'k']
for k, col in zip(unique_labels, colors):
label = f'クラスター {k}'
if k == -1:
# ノイズポイントは薄いグレーで表示
col = 'lightgray'
label += '(ノイズ)'
class_member_mask = (labels == k)
xyz = data[class_member_mask]
ax.scatter(xyz[:, 0], xyz[:, 1], xyz[:, 2], c=col, label=label)
# コアサンプルを赤でプロット
core_samples = data[core_sample_indices]
ax.scatter(core_samples[:, 0], core_samples[:, 1], core_samples[:, 2], s=100, facecolors='none', edgecolors='r', label='コアサンプル')
ax.set_title('DBSCAN 3Dクラスタリング')
ax.set_xlabel('説明変数 1')
ax.set_ylabel('説明変数 2')
ax.set_zlabel('説明変数 3')
ax.legend()
plt.show()
多次元データを次元圧縮し、クラスタリング結果を可視化
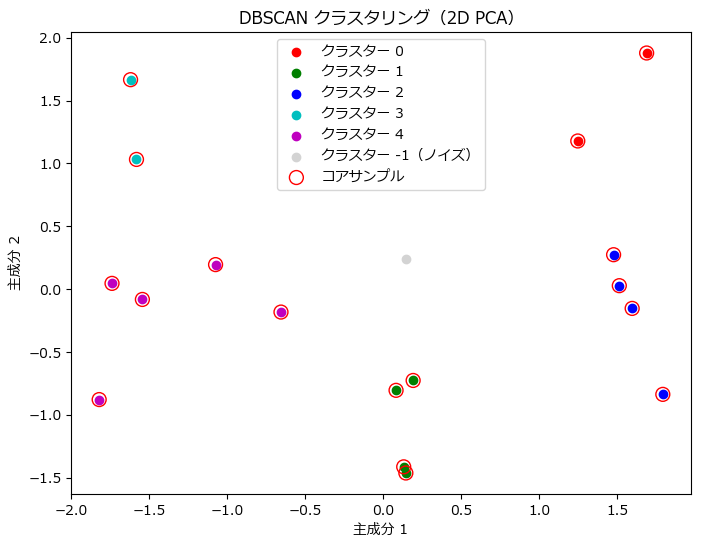
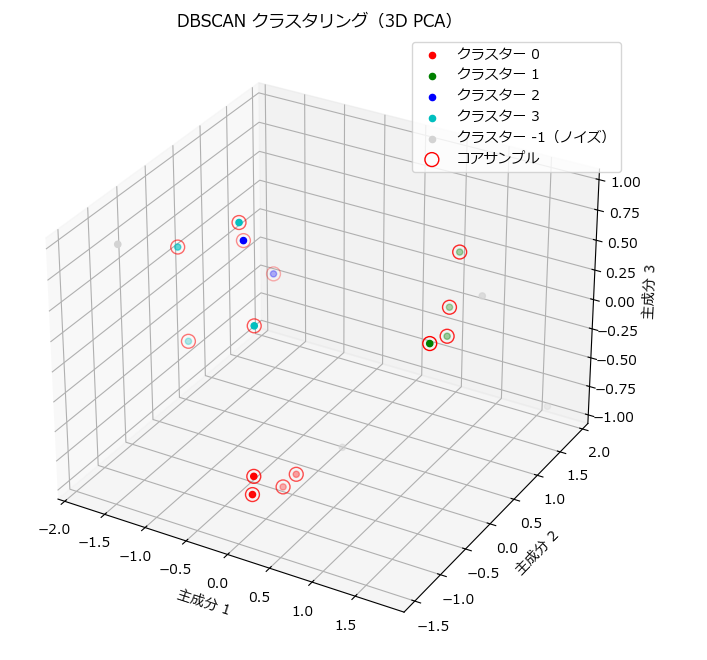
from sklearn.cluster import DBSCAN
from sklearn.decomposition import PCA
import matplotlib.pyplot as plt
from matplotlib import rcParams
import numpy as np
rcParams['font.family'] = 'Meiryo'
# 6次元データの生成
data = np.array([
[1.5, 0.7, 0.5, 1.2, 1.0, 3.6], [1.0, -1.1, 0.6, 1.2, 1.2, 1.1], [1.0, 1.1, 0.7, 1.2, 1.1, 1.3],
[1.2, 1.0, 0.5, 1.2, 1.2, 1.2], [0.7, 1.4, 0.7, 1.2, 1.3, 1.4], [1.0, -1.0, 0.6, 1.2, 1.2, 0.4],
[-1.4, -0.9, 0.6, 1.2, 1.0, 2.3], [-0.3, -0.7, 0.5, 1.2, 1.7, 1.3], [-1.4, -1.2, 0.6, 1.2, 1.0, 0.3],
[-1.3, -1.1, 0.6, 1.2, 1.1, 3.1], [0.7, 1.0, 0.5, 1.2, 1.1, 2.5], [0.8, -0.8, 0.7, 1.2, 1.3, 2.1],
[-1.4, -1.1, 0.6, 1.2, 1.2, 1.3], [1.4, 1.1, 0.7, 1.2, 1.4, 0.5], [-1.1, -0.4, 0.5, 1.2, 2.0, 1.4],
[1.1, -1.1, 0.6, 1.2, 1.1, 0.4], [-0.8, -1.5, 0.7, 1.2, 1.5, 1.5], [1.0, -0.9, 0.5, 1.2, 2.1, 1.2],
])
# --------------------------------------------------------------------------------------
# PCAを使って6次元データを2次元に変換してからDBSCANクラスタリングを実行
# --------------------------------------------------------------------------------------
# PCAによる次元削減(2次元)
pca_2d = PCA(n_components=2)
data_2d = pca_2d.fit_transform(data)
# DBSCANクラスタリングの実行(2次元)
dbscan_2d = DBSCAN(eps=0.9, min_samples=2)
labels_2d = dbscan_2d.fit_predict(data_2d)
core_sample_indices_2d = dbscan_2d.core_sample_indices_
# 2次元クラスタリング結果の可視化
plt.figure(figsize=(8, 6))
unique_labels_2d = set(labels_2d)
colors = ['r', 'g', 'b', 'c', 'm', 'y', 'k']
for k, col in zip(unique_labels_2d, colors):
label = f'クラスター {k}'
if k == -1:
col = 'lightgray'
label += '(ノイズ)'
class_member_mask = (labels_2d == k)
xy = data_2d[class_member_mask]
plt.scatter(xy[:, 0], xy[:, 1], c=[col], label=label)
# コアサンプルを赤でプロット
core_samples_2d = data_2d[core_sample_indices_2d]
plt.scatter(core_samples_2d[:, 0], core_samples_2d[:, 1], s=100, facecolors='none', edgecolors='r', label='コアサンプル')
plt.title('DBSCAN クラスタリング(2D PCA)')
plt.xlabel('主成分 1')
plt.ylabel('主成分 2')
plt.legend()
plt.show()
# --------------------------------------------------------------------------------------
# PCAを使って6次元データを3次元に変換してからDBSCANクラスタリングを実行
# --------------------------------------------------------------------------------------
# PCAによる次元削減(3次元)
pca_3d = PCA(n_components=3)
data_3d = pca_3d.fit_transform(data)
# DBSCANクラスタリングの実行(3次元)
dbscan_3d = DBSCAN(eps=0.9, min_samples=2)
labels_3d = dbscan_3d.fit_predict(data_3d)
core_sample_indices_3d = dbscan_3d.core_sample_indices_
# 3次元クラスタリング結果の可視化
fig = plt.figure(figsize=(10, 8))
ax = fig.add_subplot(111, projection='3d')
unique_labels_3d = set(labels_3d)
for k, col in zip(unique_labels_3d, colors):
label = f'クラスター {k}'
if k == -1:
col = 'lightgray'
label += '(ノイズ)'
class_member_mask = (labels_3d == k)
xyz = data_3d[class_member_mask]
ax.scatter(xyz[:, 0], xyz[:, 1], xyz[:, 2], c=[col], label=label)
# コアサンプルを赤でプロット
core_samples_3d = data_3d[core_sample_indices_3d]
ax.scatter(core_samples_3d[:, 0], core_samples_3d[:, 1], core_samples_3d[:, 2], s=100, facecolors='none', edgecolors='r', label='コアサンプル')
ax.set_title('DBSCAN クラスタリング(3D PCA)')
ax.set_xlabel('主成分 1')
ax.set_ylabel('主成分 2')
ax.set_zlabel('主成分 3')
ax.legend()
plt.show()
製造業向けのクラスタリング自作クラス
DBSCANを手軽に実行するための DBSCANUtilクラスです。
DataFrameとしてデータを用意し、クラスタリングしたいカラムをリストで指定するだけで、クラスタリングが行えます。
尚、K-Meansと異なり、DBSCAN自身が保存したモデルからの予測に対応していないため、モデルの保存と読み出しメソッドは用意していません。
DBSCANUtilクラスのインスタンスを渡すと、グラフを描画する関数も用意しています。
2次元のグラフは plot_clusters()、3次元のグラフは plot_clusters_3d()を使います。
DBSCANUtilクラスの使い方
- DBSCANでクラスタリングしたい対象データをDataFrameに格納し、
DBSCANUtilの引数にDataFrameを指定してインスタンスを生成します。 fit()メソッドの引数にカラム名のリスト(columns)を指定して呼び出します。- インスタンス作成時ではなく、
fit()の引数にDataFrameを指定することも可能です。 - クラスタリングした結果は
labelsとcore_sample_indices_のプロパティから取得可能です。
# データの生成
df = pd.DataFrame([
[1.0, 2.0, 1.5], [1.2, 3.2, 1.8], [1.8, 1.8, 1.0], [1.3, 1.3, 0.9], [0.7, 1.7, 1.2],
[5.0, 6.0, 5.5], [4.3, 6.3, 5.8], [3.7, 5.7, 5.0], [5.4, 6.4, 5.6], [4.1, 5.5, 5.3],
[9.0, 8.0, 9.5], [9.3, 9.3, 9.8], [8.2, 9.7, 8.9], [9.1, 7.4, 9.0], [8.2, 9.6, 9.3]
], columns=['Feature1', 'Feature2','Feature3'])
# モデル作成~モデル保存まで
dbs = DBSCANUtil(df)
dbs.fit(['Feature1', 'Feature2','Feature3'], eps=0.9, min_samples=2)
# 結果を表示
print(dbs.labels)
plot_clusters_3d(dbs)DBSCANユーティリティクラス
| メソッド名 | 説明 | パラメータ | 戻り値 |
|---|---|---|---|
| __init__( df, pca = 0 ) | クラスの初期化 | df: データフレーム pca: PCA次元数 | なし |
| fit( cloumns, df=None, dbscan_paramms ) | DBSCANモデルの学習 | columns: 使用するカラム df: データフレーム dbscan_paramms: DBSCANのパラメータ | DBSCANモデル |
| read_csv( file_name, encoding="shift-jis" ) | CSVファイルからデータを読み込む | file_name: ファイルパス encoding: エンコーディング | なし |
import pandas as pd
from sklearn.cluster import DBSCAN
from sklearn.decomposition import PCA
class DBSCANUtil:
def __init__(self, df=None, pca=0):
"""
DBSCANUtilクラスの初期化メソッド
:param df: 初期データフレーム (pd.DataFrame)
:param pca: PCAを適用する次元数 (0: 使用しない, 2: 2次元, 3: 3次元)
"""
self.df = None if df is None else df.copy() # データフレームのコピーを保持
self.model = None
self.labels = []
self.core_sample_indices_ = []
self.components_ = []
self.columns = []
self.pca = pca # PCA次元数の指定
def fit(self, columns: list, df: pd.DataFrame = None, **dbscan_params):
if df is not None:
self.df = df
self.columns = columns
X = self.df[columns].values
if self.pca > 0:
# PCAによる次元削減
pca_model = PCA(n_components=self.pca)
X = pca_model.fit_transform(X)
# DBSCANモデルを作成
self.model = DBSCAN(**dbscan_params)
self.model.fit(X) # クラスタリングを実行
self.labels = self.model.labels_ # ラベルを取得
self.core_sample_indices_ = self.model.core_sample_indices_
self.components_ = self.model.components_
return self.model
def read_csv(self, file_name, encoding="shift-jis"):
"""
CSVファイルからデータを読み込むメソッド
:param file_name: 読み込むファイルのパス (str)
:param encoding: ファイルのエンコーディング (str)
"""
self.df = pd.read_csv(file_name, encoding=encoding) # データフレームに読み込みグラフ描画関数
| 関数名 | 説明 | 引数 | 戻り値 | 備考 |
|---|---|---|---|---|
| plot_clusters( dbscan_util, x_col=None, y_col=None ) | 2次元クラスタリングの結果を可視化する関数です。クラスタの中心と各データ点をプロットします。 | dbscan_util: DBSCANUtilのインスタンス x_col: x軸にプロットするカラム名 y_col: y軸にプロットするカラム名 | None | x_col, y_colが指定されない場合は、DBSCANUtil.columnsの最初の2つが使用されます。 |
| plot_clusters_3d( dbscan_util, x_col=None, y_col=None, z_col=None ) | 3次元クラスタリングの結果を可視化する関数です。クラスタの中心と各データ点をプロットします。 | dbscan_util: DBSCANUtilのインスタンス x_col: x軸にプロットするカラム名 y_col: y軸にプロットするカラム名 z_col: z軸にプロットするカラム名 | None | x_col, y_col, z_colが指定されない場合は、DBSCANUtil.columnsの最初の3つが使用されます。 |
下記は2次元グラフの描画関数です。
import numpy as np
import matplotlib.pyplot as plt
from matplotlib import rcParams
rcParams['font.family'] = 'Meiryo'
def plot_clusters(dbscan_util, x_col=None, y_col=None):
"""
クラスタリング結果を可視化
- dbscan_util: DBSCANUtilのインスタンス
- x_col, y_col: プロットするカラム名(省略時にはdbscan_util.columnsを使用)
"""
if not isinstance(dbscan_util, DBSCANUtil):
raise ValueError("引数はDBSCANUtilのインスタンスでなければなりません。")
# x_col, y_colが指定されていない場合、dbscan_util.columnsを使用
if x_col is None or y_col is None:
if len(dbscan_util.columns) < 2:
raise ValueError("x_col と y_col が指定されていない場合、columnsに2つ以上のカラムが必要です。")
x_col = dbscan_util.columns[0] # columnsの1番目をx軸
y_col = dbscan_util.columns[1] # columnsの2番目をy軸
plt.figure(figsize=(8, 6))
# クラスタごとにデータポイントをプロット
unique_labels = np.unique(dbscan_util.labels)
for cluster in unique_labels:
cluster_data = dbscan_util.df[dbscan_util.labels == cluster]
col = 'lightgray' if cluster == -1 else None
plt.scatter(cluster_data[x_col], cluster_data[y_col], label=f"クラスター {cluster}(ノイズ)" if cluster == -1 else f"クラスター {cluster}", c=col)
# コアサンプルを赤でプロット
core_samples = dbscan_util.df.iloc[dbscan_util.model.core_sample_indices_]
plt.scatter(core_samples[x_col], core_samples[y_col], s=100, facecolors='none', edgecolors='r', label='コアサンプル')
plt.xlabel(x_col)
plt.ylabel(y_col)
plt.legend()
plt.title("DBSCAN クラスタリング結果の可視化")
plt.show()下記は3次元グラフの描画関数です。
import numpy as np
import matplotlib.pyplot as plt
from matplotlib import rcParams
rcParams['font.family'] = 'Meiryo'
def plot_clusters_3d(dbscan_util, x_col=None, y_col=None, z_col=None):
"""
3Dクラスタリング結果を可視化
- dbscan_util: DBSCANUtilのインスタンス
- x_col, y_col, z_col: プロットするカラム名(省略時にはdbscan_util.columnsを使用)
"""
if not isinstance(dbscan_util, DBSCANUtil):
raise ValueError("引数はDBSCANUtilのインスタンスでなければなりません。")
# x_col, y_col, z_colが指定されていない場合、dbscan_util.columnsを使用
if x_col is None or y_col is None or z_col is None:
if len(dbscan_util.columns) < 3:
raise ValueError("x_col, y_col, z_col が指定されていない場合、columnsに3つ以上のカラムが必要です。")
x_col = dbscan_util.columns[0] # columnsの1番目をx軸
y_col = dbscan_util.columns[1] # columnsの2番目をy軸
z_col = dbscan_util.columns[2] # columnsの3番目をz軸
fig = plt.figure(figsize=(10, 8))
ax = fig.add_subplot(111, projection='3d')
# クラスタごとにデータポイントをプロット
unique_labels = np.unique(dbscan_util.labels)
for cluster in unique_labels:
cluster_data = dbscan_util.df[dbscan_util.labels == cluster]
col = 'lightgray' if cluster == -1 else None
ax.scatter(cluster_data[x_col], cluster_data[y_col], cluster_data[z_col], label=f"クラスター {cluster}(ノイズ)" if cluster == -1 else f"クラスター {cluster}", c=col)
# コアサンプルを赤でプロット
core_samples = dbscan_util.df.iloc[dbscan_util.model.core_sample_indices_]
ax.scatter(core_samples[x_col], core_samples[y_col], core_samples[z_col], s=100, facecolors='none', edgecolors='r', label='コアサンプル')
ax.set_xlabel(x_col)
ax.set_ylabel(y_col)
ax.set_zlabel(z_col)
ax.legend()
ax.set_title("3D DBSCAN クラスタリング結果の可視化")
plt.show()
まとめ
本記事では、DBSCANを活用したクラスタリングの基本から応用、製造業での実践的な活用例までを解説しました。
DBSCANは密度に基づいたクラスタリング手法として、製造現場のデータ解析や異常検知で非常に有効です。その柔軟性とノイズ耐性により、複雑なパターンの発見や外れ値の識別が可能となり、製品品質の向上や設備保全の効率化につながります。
一方で、パラメータ設定やデータ規模の課題を理解し、適切に対処することが成功の鍵となります。
本記事の内容が、皆さんのデータ分析に役立つことを願っています。ぜひ実践してみてください。






コメント